Python pour les scientifiques
Une rapide introduction
CentraleSupélec
Introduction
Syllabus
Module SPM-INF-002:
- 2 CM : calcul scientifique, gestion de données et visualisation
- 6 TPs disponibles sur https://jeremyfix.github.io/Python-for-scientists/ :
- TP1 : manipulation de tableaux, calculs matriciels
- TP2 : Interpolation et optimisation
- TP3 : Traitement du signal
- TP4 : Analyse de données avec pandas
- TP5 : Statistiques
- TP6 : Apprentissage automatique avec scikit-learn
Evaluation par les rendus des TPs, limite 1 semaine après le TP.
Ressources
Scientific python lectures aborde :
- le langage python
- Numpy, Matplotlib, Scipy,
- des sujets avancés (interface python/C++)
- …

- Python pour la datascience : numpy, pandas
- Dive into python3,
- www.pythoncheatsheet.org
Petit tour d’horizon
L’écosystème python est très riche en librairies scientifiques :
- Numpy pour le calcul,
- Pandas pour l’analyse de données,
- Scipy pour le traitement du signal,
- Scikit-learn pour l’apprentissage automatique,
- Matplotlib/Seaborn pour les tracés,
- Sympy pour le calcul symbolique,
Introduction à l’interpréteur python
Les interpréteurs
Python est un langage interprété
- Langage compilé = déjà transformé en instructions CPU
- Langage interprété = un programme (compilé :) ) transforme “à la volée” votre code en instructions.
“à la volée”: cpython peut créer un fichier .pyc qui contient le code compilé (bytecode).
Les interpréteurs
- Le plus connu, de l’inventeur du langage, Guido Van Rossum : CPython
- Mais aussi IronPython, Jython, Pypy, Pyston, RustPython, Micro Python
Performances de différentes version du “même” code
Un code lent et gourmand en mémoire
Mauvais code
rangeexplicité en mémoire \(\neq\) paresseuse- boucle
foren python - l’accès indicé
l[i]pourrait être problématique mais les listes python sont des tableaux et pas des listes chaînées
Un peu plus rapide, moins gourmand en mémoire
Mauvais code
- la boucle for est exécutée en python, c’est à dire interprétée
Le “même” code mais plus efficace
Bien plus vite tout en restant paresseux en mémoire
Fondamentalement différent
- Appel de la fonction
built-insum, déjà compilée
Plus rapide mais plus gourmand avec numpy
Un autre bénéfice de la compilation : le code vectorisé
Instructions vectorielles
- les processeurs modernes disposent d’instructions vectorielles, e.g. AVX
- une instruction vectorielle (SIMD : Single Instruction Multiple Data) peut appliquer la même opération sur plusieurs données (e.g. \(4\) données)
- l’interpréteur python
CPythonne supporte pas les instructions vectorisées, - les compilateurs, e.g. pour C++ comme
gccouclang, réorganisent le code pour exploiter les instructions SIMD - les outils/librairies comme numpy, cython ou pypy compilent du code python et peuvent utiliser les instructions SIMD
Installation des packages
Environnement virtuel
Un environnement virtuel est :
- un répertoire isolé d’installation
- qui a vocation à ne contenir que les dépendances de votre projet en cours
- dont la construction est documentée
Il permet la reproductibilité (Benureau and Rougier 2018), avec le contrôle des packages installés et de leurs versions.
Différentes options :
venv(inclus dans Python depuis la version 3.3) : python package manager, ne permet pas de changer de version de pythonanaconda,miniconda,mamba,micromamba, etc… : system package manager. Permet de changer de version de python, installer des dépendances autres que des packages python (e.g. C++, Java, Rust, …), mais parfois un peu lourduvhttps://docs.astral.sh/uv : python package manager, flexible sur la version de python, ne permet d’installer que des paquets python
Exemple avec micromamba
Pour installer micromamba (en utilisateur normal):
Pour construire un environnement virtual local dédié :
Pour installer des dépendances :
On peut exporter, recharger, etc.. un environnement.
Exemple avec uv
Pour installer uv (en utilisateur normal) :
Pour construire un environnement virtuel local dédié :
Pour installer des dépendances :
Environnement de développement
VS Code
Warning
Vous devez utiliser un Integrated Development Environment (IDE)
Suggestion : utilisez Visual Studio Code + tout son environnement de plugins qui permet :
- de gérer votre projet (création, organisation des fichiers)
- disposer de la coloration syntaxique, complétion de code, etc…
- mettre en place un environnement virtuel et exécuter le code dans cet environnement
- utiliser des notebooks jupyter
- interagir avec git
VS Code https://code.visualstudio.com/docs/languages/python
=> Démo avec Venv
Jupyter notebook/lab
On peut également travailler avec des notebooks jupyter.
- installable localement ou en ligne (https://mybinder.org/, google colab)
Fonctionnalités :
- supporte des noyaux python, c++, R, …
- permet de combiner du texte et du rendu
- permet d’inclure des widgets dynamiques (e.g. sliders)
Voir ces exemples à essayer dans le navigateur, e.g. le simulateur de l’attracteur de Lorentz
Python : bases
Rappels de syntaxe de base
- ATTENTION à l’indentation
- types
int,float,bool,set - types “séquence” immutables:
str,tuple - type “séquence” mutable :
list - type séquence clé/valeur :
dict - interroger le type
type(x), tester le typeisinstance(x, letype), connaître les méthodes applicablesdir(x)
- opérateur d’affectation
x = 3, walrus
- opérateurs arithmétiques (+, -, /, *), et logiques (and, or, not)
Rappels de syntaxe de base
Sur les listes
Faire attention aux constructions du type :
Complexité en temps en \(O(|L|^2)\) pour une liste chaînée. Complexité en temps en \(O(|L|)\) pour un tableau.
Rappels de syntaxe de base
Les générateurs
Une construction de liste par compréhension est exécutée à la déclaration.
Pour économiser de la mémoire (e.g. permettant de représenter d’énormes structures), on peut utiliser des générateurs
Rappels de syntaxe de base
Sur les fonctions
Définition d’une fonction : def fname():
On peut utiliser un nombre variable d’arguments positionnels *args et/ou nommés **kwargs (kwargs = keyword arguments). Voir Python args and kwargs.
Les lambda fonctions allègent la déclaration des fonctions :
Modificateurs de fonctions : les décorateurs
Voir https://www.pythoncheatsheet.org/cheatsheet/decorators
@CountCallNumber
def say_hi(name"):
print("Hi ! My Name is "+ name)
class CountCallNumber:
def __init__(self, func):
self.func = func
self.call_number = 0
def __call__(self, *args, **kwargs):
self.call_number += 1
print("This is execution number " + str(self.call_number))
return self.func(*args, **kwargs)Rappels de syntaxe de base
Loguer avec print: Pour déboguer un programme, on utilise bien souvent des print(...)
Que l’on peut combiner avec des f-strings :
Que l’on peut combiner avec des options de formattage :
Loguer avec logging : On peut aussi utiliser des loggers du module logging :
import logging
logging.basicConfig(format="%(levelname)s - %(asctime)s: %(message)s",
datefmt= "%d/%m/%y %H:%M:%S", level=logging.INFO)
logging.info("Une info")
logging.warning("Un warning")
logging.error("Une erreur")
logging.basicConfig(filename="monlog.log",
format="%(levelname)s - %(asctime)s: %(message)s",
datefmt= "%d/%m/%y %H:%M:%S", level=logging.INFO)Lire/ecrire dans un fichier
De manière générale sur un texte en ASCII :
Lecture, avec un with statement:
Ecriture :
ATTENTION C’est une très mauvaise idée de stocker en ASCII des données numériques.
La valeur “1.23439949959” en ASCII occupe \(13 \times 8 = 104 bits\). Alors qu’un flottant peut occuper \(64\), \(32\), \(16\) bits.
Si ce sont des tableaux de valeurs, utilisez numpy.savez.
Vous pouvez aussi construire votre propre format de fichier et utiliser fh.seek(), fh.read(num_bytes), struct.pack(), struct.unpack(). Voir [struct](https://docs.python.org/3/library/struct.html]
Sur des formats spécifiques yaml, xml, json, utilisez des parsers écrit pour le yaml, xml, json
Python: classes, threads, UI, packing
Un problème pour se motiver
Problème Simuler un système d’Ising (Sethna (2006), Hayes (2000)). Moment magnétique des atomes arrangés sur une grille régulière \(\sigma_i \{-1, 1\}\). Pour une grille finie, il existe un nombre fini de configurations mais avec des dynamiques intriguantes.
La probabilité d’occuper un état du système \(\sigma\) est \(P(\sigma) = \frac{1}{Z}\exp(-\beta E(\sigma))\), avec
\[ E(\sigma) = -\sum_{i < j} J_{i,j} \sigma_i \sigma_j \]
- transition déterministe si l’énergie globale diminue, \(\Delta E < 0\),
- transition stochastique si l’énergie augmente \(\Delta E > 0\), avec probabilité \(\exp(-\beta \Delta E) \in [0, 1]\)
Il existe des transitions de phase, en particulier pour \(\beta \approx 2.269\).
Voir la description complète du système sur https://tutos.metz.centralesupelec.fr/TPs/Intro-Python/index.html#an-example-project-simulating-the-ising-model
Les classes
Concept fondamental de programmation orientée objet (OOP).
- Un objet = des attributs modifiables par des méthodes qui forment un tout cohérent. On parle aussi de classe,
- Une réalisation d’un objet est appelée une instance,
- peut être organisé de manière hiérarchique,
- peut redéfinir/spécifier des opérations
On peut construire une instance d’un objet :
On dispose également de beaucoup de méthodes spéciales : __lt__, __add__, __str__, …
On peut construire une hiérarchie de classes, on doit alors appeler le constructeur de la classe mère.
Illustrations de la programmation orientée objet
Problème Écrire un évaluateur d’expressions mathématiques.
class BinaryOp(Expression):
def __init__(self, fun, left, right):
self.fun = fun
self.left, self.right = left, right
def __call__(self, env):
return self.fun(self.left(env), self.right(env))
class Add(BinaryOp):
def __init__(self, left, right):
super().__init__(lambda x, y: x+y, left, right)
def __str__(self):
return f"({self.left} + {self.right})"
class Multiply(BinaryOp):
def __init__(self, left, right):
super().__init__(lambda x, y: x*y, left, right)
def __str__(self):
return f"({self.left} * {self.right})"Exemple
Les classes
On décompose le projet en \(3\) classes :
- un simulateur qui s’occupe de la physique
- une interface en ligne de commande
Console - une interface graphique
IsingGUI
Les threads ?
Un programme peut déclencher plusieurs fils d’exécutions, les threads. Ici :
- un thread pour le simulateur
- un thread pour l’interaction avec l’utilisateur (le principal)
Cela permet à la simulation de continuer à tourner même si l’utilisateur n’interagit pas.
import threading
class Simulator(threading.Thread):
def __init__(self, lattice_size, kT):
threading.Thread.__init__(self)
self.lock = threading.RLock()
self.spins = np.random.random((lattice_size, lattice_size)) > 0.5
def run(self):
while self.is_kept_alive():
if self.running:
self.step()
def stop(self):
with self.lock:
self.keeps_alive = False
def get_spins(self):
with self.lock:
return self.spins.copy()Au lieu des threads, on aurait pu utiliser du multiprocessing.
Installation et exécution
Installation (grâce au setup.py)
Exécution
Packaging du projet
Le projet est packagé avec setuptools.
La python packaging authority (pypa) maintient un tutorial sur le packaging, voir https://setuptools.pypa.io/en/latest/userguide/quickstart.html
Le packaging peut se faire avec un setup.py :
import setuptools
with open("README.md", "r") as fh:
long_description = fh.read()
setuptools.setup(
name="Ising",
version="1.0",
author="CentraleSupelec",
author_email="Jeremy.Fixcentralesupelec.fr",
description='A simulator of the Ising model with '
'the Metropolis algorithm',
long_description=long_description,
long_description_content_type="text/markdown",
url='http://tutos.metz.centralesupelec.fr/Intro_Python',
packages=setuptools.find_packages(),
classifiers=[
"Programming Language :: Python :: 3",
"License :: OSI Approved :: MIT License",
"Operating System :: OS Independent",
],
python_requires='>=3.6',
install_requires=['numpy', 'scipy', 'PyQt5', 'matplotlib', 'opencv-python-headless']
)Mais pourrait aussi utiliser pyproject.toml.
Et vous pourriez déposer votre librairie sur pypi, ou conda.
Calcul scientifique avec Numpy
Introduction
Numpy : Numerical Python
- Librairie C++ avec un wrapper python,
- qui offre des tableaux multi-dimensionnels et des opérations optimisées
NumPy is the fundamental package for scientific computing in Python. It is a Python library that provides a multidimensional array object, various derived objects (such as masked arrays and matrices), and an assortment of routines for fast operations on arrays, including mathematical, logical, shape manipulation, sorting, selecting, I/O, discrete Fourier transforms, basic linear algebra, basic statistical operations, random simulation and much more.- Documentation : https://numpy.org/doc/stable/,
- Tutoriels : https://lectures.scientific-python.org/intro/numpy/index.html et sur les Quickstart sur la page de documentation,
- Le “Try it in your browser!”, voir par exemple le bas de https://numpy.org/doc/2.3/reference/generated/numpy.linalg.inv.html
- Code source : https://github.com/numpy/numpy
Création de tableaux
Plusieurs approches pour créer un tableau Numpy :
- à partir d’une liste (éventuellement de listes de listes de ..)
- à partir de fonctions dédiées :
np.zeros,np.ones,np.arange,np.linspace,np.random.rand, etc. - à partir de fichiers :
np.loadtxt,np.genfromtxt,np.load, etc.
# Tableau 1D de taille (5, )
np.array([1, 2, 3, 4, 5])
# Tableau 2D de taille (2, 3)
np.array([[1, 2, 3], [4, 5, 6]])
# Tableau 2D de taille (3, 4) rempli de zéros
np.zeros((3, 4))
# Tableau 6D de taille (2, 3, 4, 5, 6, 7) rempli de uns
np.ones((2, 3, 4, 5, 6, 7))
# Tableau 1D de 0 à 10 par pas de 2
np.arange(0, 10, 2)
# Tableau 1D de taille (5, ) avec des valeurs entre 0 et 1
np.linspace(0, 1, 5)
# Tableau 2D de taille (3,3) avec des valeurs aléatoires entre 0 et 1
np.random.rand(3, 3)Propriétés des tableaux
Les tableaux ont :
- une forme (shape) : nombre de dimensions et taille dans chaque dimension,
- un type (dtype) : type des éléments du tableau,
- un nombre d’éléments (size) : nombre total d’éléments dans le tableau,
- un nombre de dimensions (ndim) : nombre de dimensions du tableau.
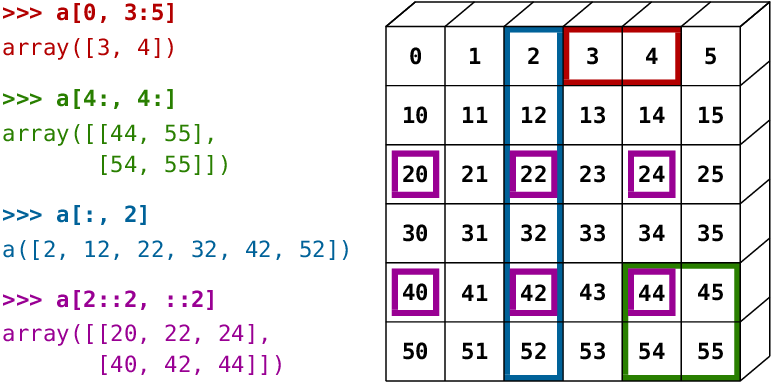
Accès aux éléments
On accède aux éléments :
- par index,
- par slicing, en utilisant la syntaxe python
start:stop:step, - par masquage booléen
Pour l’efficacité en mémoire et temps:
- le slicing crée une vue sur le tableau original, pas une copie.
- Mais, le fancy indexing crée une copie, pas une vue.
On peut le vérifier avec l’attribut base
a = np.array([[1, 2, 3], [4, 5, 6]])
# Accès par index
# Premier élément de la première ligne
a[0, 0] # 1
# Accès par slicing
# Deuxième ligne
# Equivalent à a[1]
a[1, :] # [4 5 6]
# Première colonne
a[:, 0] # [1 4]
# Toutes les lignes, colonnes d'indice pair
a[:, ::2] #
a[:, ::2] = 0 # Modifie a
# Masquage booléen : fancy indexing
mask = a > 3 # np.array de dtype bool
b = a[mask] # [4 5 6]
b[0] = 10 # Ne Modifie pas aAccès aux éléments (suite)
Ne jamais faire
mais utiliser l’ellipsis :
Manipulations sur la forme des tableaux
On peut modifier la forme d’un tableau par :
np.reshape: retourne une vue du tableau avec une nouvelle forme,np.flattenounp.ravel: retourne une copie ou une vue du tableau aplati (1D),np.transpose: retourne une vue du tableau transposé,np.pad: ajoute une bordure autour du tableau.np.expand_dims,x = x[np.newaxis, :]: ajoute une nouvelle dimension.
On peut combiner/diviser plusieurs tableaux par :
np.concatenate: concatène plusieurs tableaux le long d’un axe,np.stack: empile plusieurs tableaux le long d’un nouvel axe,np.hstacketnp.vstack: concatène horizontalement ou verticalement plusieurs tableaux.np.split,np.hsplit,np.vsplit: divise un tableau en plusieurs sous-tableaux.
On peut permuter les éléments d’un tableau par :
np.transpose,np.swapaxesnp.roll
Opérations arithmétiques sur les tableaux
Les opérations arithmétiques sont appliquées élément par élément +, ‘/’, -, *. Il existe également les opérations logiques.
a = np.array([[1, 2, 3], [4, 5, 6]])
b = np.array([[10, 20, 30], [40, 50, 60]])
# Addition
c = a + b # [[11 22 33] [44 55 66]]
# Soustraction
d = b - a # [[9 18 27] [36 45 54]]
# Multiplication terme à terme
e = a * b # [[10 40 90] [160 250 360]]
# Division terme à terme
f = b / a # [[10. 10. 10.] [10. 10. 10.]]Attention : * est le produit terme à terme, pas le produit matriciel. Le produit matriciel est obtenu par np.matmul ou simplement @
Illustration - Système de GrayScott
Problème Simuler le système de réaction-diffusion de GrayScott dont l’évolution des champs spatialisés \(u(x,t)\), \(v(x,t)\), \(x\in \mathbb{R}^2\) est régie par :
\[ \begin{eqnarray*} \frac{\partial u}{dt}(x,t) &=& D_u \nabla^2 u(x,t) - u(x,t) v^2(x,t) \\ & &+ F(1-u(x,t))\\ \frac{\partial v}{dt}(x,t) &=& D_u \nabla^2 v(x,t) + u(x,t) v^2(x,t) \\ && - (F+k) v(x,t) \end{eqnarray*} \]
def step(ut_1, vt_1, Du, Dv, F, k):
uvv = ut_1 * vt_1**2
lu = laplacian(ut_1)
lv = laplacian(vt_1)
ut[...] = ut_1 + dt * (Du * lu - uvv + F*(1-ut_1))
vt[...] = vt_1 + dt * (Dv * lv + uvv - (F + k) * vt_1)
def laplacian(src):
"""
u is a 2D nd array
"""
padded_src = np.pad(src, pad_width=1, mode="constant") # zero padding src
laplacian = (
0
+ padded_src[0:-2, 1:-1]
+ 0
+ padded_src[1:-1, 0:-2]
- 4 * padded_src[1:-1, 1:-1]
+ padded_src[1:-1, 2:]
+ 0
+ padded_src[2:, 1:-1]
+ 0
)
return laplacian[1:-1, 1:-1]avec \(\nabla^2\) le laplacien discret :
\[ \nabla^2 u(x,y) = \frac{u(x-\delta, y) + u(x+\delta, y) + u(x, y-\delta) + u(x, y+\delta) - 4 u(x,y)}{\delta^2} \]
Illustration - Système de Grayscott
Fonctions Mathématiques
Les fonctions Mathématiques s’appliquent sur tout les éléments, e.g. np.exp, np.sin, np.log, etc mais également les puissances.
e.g. Calcul d’une gaussienne centrée en \(x=2\) avec écart-type \(\sigma=0.5\)
On peut également calculer (selon un axis=..):
- la somme :
np.sum, - le plus petit/grand :
np.min,np.max,np.argmin,np.argmax, - la moyenne :
np.mean\(\frac{1}{N} \sum_i X[..., i, ...]\), l’écart typenp.std\(\sqrt{\frac{1}{N} \sum_i (X[..., i, ...] - \mu_i)^2}\) - la covariance :
np.cov\(\sum_i (x_i - \mu)^T (x_i - \mu)\) - …
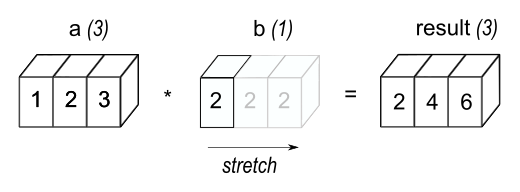
Broadcasting
Le broadcasting est un mécanisme qui permet d’effectuer des opérations entre des tableaux de formes différentes.
Numpy “étend” automatiquement les dimensions des tableaux pour qu’ils soient compatibles pour l’opération.
De la documentation sur le broadcasting
La règle : des opérations sur deux tableaux sont applicables si les dimensions sont compatibles.
Les dimensions sont compatibles si, 1) les tableaux ont le même nombre de dimensions, 2) partant de la droite (des dimensions)
- les dimensions sont égales
- ou sont différentes mais l’une d’elle est égale à \(1\) \(\rightarrow\) Broadcast
Sinon vous obtiendrez l’exception ValueError: operands could not be broadcast together
Broadcasting - example
Par exemple, on souhaite calculer la valeur d’une fonction gaussienne \(f(x)\) centrée en \(\mu\in\mathbb{R}^2\), sur une grille régulière centrée sur \(\mu\) :
\[ \forall x \in \mathbb{R}^2, f(x) = \frac{1}{\sigma \sqrt{2\pi}} \exp(\frac{- \|x - \mu\|_2^2}{2\sigma^2}) \]
if __name__ == '__main__':
mu = np.array([1, 2])
sigma = 0.3
step = 0.1
Nsteps = 10
X = gaussian(mu, sigma, step, Nsteps)
def gaussian(mu: np.ndarray, sigma: float, step: float, Nsteps: int) -> np.ndarray:
# Generate the regular grid
# (-N, -(N-1), ..., -1, 0, 1, ..., N-1, N) * step
intsteps = (np.arange(Nsteps) + 1)
deltasteps = np.concatenate([-intsteps[::-1], [0], intsteps]) * step
# Generate the steps centered on mu
# mu is (2, ), we use broadcasting on mu and deltasteps to generate
# mu (2, 1) + deltasteps(1, M) => mu + deltasteps is (2, M)
mu_delta = mu[:, np.newaxis] + deltasteps[np.newaxis, :]
# Generate the meshgrid with this values
# X and Y are of shape (2*Nsteps + 1, 2*Nsteps + 1)
X, Y = np.meshgrid(mu_delta[0], mu_delta[1])
# Concatenate the X, Y matrices
# xy is (( (2*Nsteps + 1) * (2*NSteps + 1), 2)
xy = np.concatenate((X.ravel()[:,np.newaxis], Y.ravel()[:, np.newaxis]), axis=1)
# Compute tha gausian values
# To compute the distance between the grid points is computed between xy and mu
# To broadcast, we do : xy (M, 2) - mu(1, 2)
dist = ((xy - mu[np.newaxis, :])**2).sum(axis=1)
values = 1.0 / (2.0 * sigma * np.sqrt(2.0 * np.pi)) * np.exp(- dist / (2.0 * sigma**2))
# Values is of shape (( (2*Nsteps + 1) * (2*NSteps + 1), ) that we reshape as
# (2*Nsteps + 1, 2*Nsteps + 1)
values = values.reshape((2*Nsteps + 1, 2*Nsteps + 1))
return values
Algèbre linéaire
Numpy supporte bien évidemment toutes les routines utiles en algèbre linéaire, dans le sous-package np.linalg:
- produit matriciel , matrice-vector :
np.dot - calcul de la trace
np.linalg.trace, - calcul de l’inverse :
np.linalg.inv, du déterminantnp.linalg.det - calcul des normes :
np.norm- norme \(p\) pour un vecteur \((\sum_i x_i^p)^{1/p}\), Frobenius pour une matrice, - diagonalisation, valeurs propres, vecteurs propres :
np.linalg.eig - décompositions de Cholesky
np.linalg.cholesky\(X = L L^T\) (L triangulaire inférieure), SVDnp.linalg.svd\(X = U S V^T\)
Algèbre linéaire - illustrations
Moindres carrés régularisés Soit \(n, m \in \mathbb{N}\), \(A\in \mathbb{R}^{n,m}\), \(b\in \mathbb{R}^{n}\), trouver \(x \in \mathbb{R}^m\) qui minimise :
\[ argmin_x J(x) = \|A x - b\|_2^2 = \sum_{i=0}^{n-1} (\sum_{j=0}^{m-1} a_{i,j}x_j - b_i)^2 \]
En fonction du range de \(A\), il peut exister une infinité de solution. Si \(A\) est singulière, une solution est obtenue en résolvant le problème des moindres carrés régularisés \(argmin_x J(x) + \lambda \|x\|_2^2\) , \(\lambda \in \mathbb{R}^+\) :
\[ x = (A^T A + \lambda \mathbf{I})^{-1} A^T b \]
Avec numpy :
import numpy as np
def rls(A: np.ndarray, b: np.ndarray, lbd: float) -> np.ndarray:
n, m = A.shape
inv = np.linalg.inv(A.T@A + lbd * np.eye(m))
x = np.dot(inv@A.T, b)
return x
if __name__ == '__main__':
n, m = 3, 4
A, b = np.random.random((n, m)), np.random.random((n,))
lbd = 0.001
x = rls(A, b, lbd)
print(x)Algèbre linéaire - PCA
Analyse en composantes principales Soit \(N\) vecteurs \(x_i \in \mathbb{R}^d\), trouver le vecteur \(w_0 \in \mathbb{R}^d\) et \(r\) vecteurs de projections \(w_j\) qui minimisent l’erreur de reconstruction :
\[ \begin{align} \min_{\{w_0, w_1, ..w_r\} \in \mathbb{R}^d} \sum_{i=0}^{N-1} \left|x_i - (w_0 + \sum_{j=1}^r (w_j^T (x_i-w_0))w_j )\right|_2^2 \end{align} \]
sujet à la contrainte \(\forall i, j \geq 1, w_i^T w_j = \delta_{i,j}\).
Algèbre linéaire - PCA
Algorithme:
- Centrer vos données \(\widetilde{x}_i = x_i - \bar{x}\)
- Construire la matrice \(\widetilde{\mathbf{X}} = \begin{bmatrix} \widetilde{x}_0^T \\ \dots \\ \widetilde{x}_{N-1}^T\end{bmatrix}\)
- Calculer les \(r\) vecteurs propres normalisés associés aux plus grandes valeurs propres de \(\widetilde{\mathbf{X}}^T\widetilde{\mathbf{X}}\)
Implémentation naïve https://github.com/rougier/ML-Recipes
class PCA:
def fit(self, X):
'''
Performs the PCA of X and stores the principal components.
The datapoints are supposed to be stored in the row vectors of X.
It keeps only the n_components projection vectors, associated
with the n_components largest eigenvalues of the covariance matrix X.T X
'''
# Center the datapoints
self.centroid = np.mean(X, axis=0)
# Computes the covariance matrix
sigma = np.dot((X - self.centroid).T, X - self.centroid)
# Compute the eigenvalue/eigenvector decomposition of sigma
eigvals, eigvecs = np.linalg.eigh(sigma)
# Note :The eigenvalues returned by eigh are ordered in ascending order.
# Stores the n_components eigenvectors/eigenvalues associated
# with the largest eigen values
#self.eigvals = eigvals[-self.n_components:]
#self.eigvecs = eigvecs[:, -self.n_components:]
# Stores all the eigenvectors/eigenvalues
# Useful for later computing some statistics of the variance we keep
self.eigvals = eigvals
self.eigvecs = eigvecs
def transform(self, Z):
'''
Uses a fitted PCA to transform the row vectors of Z
Remember the eigen vectors are ordered by ascending order
Denoting Z_trans = transform(Z),
The first component is Z_trans[:, -1]
The second component is Z_trans[:, -2]
...
'''
return np.dot(Z - self.centroid, self.eigvecs[:, -self.n_components:])Analyse de données avec pandas
Introduction
La librairie pandas est développée pour l’analyse de données.
Site du projet https://pandas.pydata.org/
Cheat sheet, Data Wrangling with pandas
Les très bons Getting started tutorials
Autres ressources : Workshop pandas de Stéfanie Molin et son livre Hands-On Data Analysis with Pandas: A Python data science handbook for data collection, wrangling, analysis, and visualization
Installation
Avec uv :
Avec pip :
Beaucoup d’exemples par la suite viennent du guide utilisateur Pandas.
Series et DataFrame
- une série (pd.Series) est une collection de valeurs indexées :
une série peut être indexée, slicée, etc.. comme un tableau numpy avec
iloc, et comme un dictionnaire avec[..]ouloc()une DataFrame est une collection de séries de données (comme une base de données)
d = {
"one": pd.Series([1.0, 2.0, 3.0], index=["a", "b", "c"]),
"two": pd.Series([1.0, 2.0, 3.0, 4.0], index=["a", "b", "c", "d"]),
"three": pd.Series([1.0, 2.0 ], index=["b", "d"])
}
df = pd.DataFrame(d)
print(df)
one two three
a 1.0 1.0 NaN
b 2.0 2.0 1.0
c 3.0 3.0 NaN
d NaN 4.0 2.0
df.loc['a',['one', 'three']] # pd.Series- on accède aux colonnes
df.columns, à leurs typesdf.dtypes, à l’indexdf.index - on peut ajouter (comme un dictionnaire) des colonnes
df["four"] = df["one"] + df["two"], supprimerdel df["one"]
Créer une dataframe et obtenir des infos
On peut construire une DataFrame à partir de dictionnaires, tableaux numpy, … ou de fichiers :
# Dictionnaire des colonnes
d = {
"one": pd.Series([1.0, 2.0, 3.0], index=["a", "b", "c"]),
"two": pd.Series([1.0, 2.0, 3.0, 4.0], index=["a", "b", "c", "d"]),
"three": pd.Series([1.0, 2.0 ], index=["b", "d"])
}
df = pd.DataFrame(d)
# Liste des lignes
df = pd.DataFrame([{"one": 1, "two": 2}, {"one": 5, "two": 10, "three": 20}])
# Fichier CSV
df = pd.read_csv("fichier.csv", delimiter=";")- on obtient des infos avec
df.info(),df.describe(),df.head() - statistiques descriptives (mean, std, count)
df.describe(), (unique, top, freq)df.describe(include='category')
Vérifier et corriger les types
- Les types des colonnes peuvent être le type générique “object”.
- si des données sont des temps (
datetime), des données catégoriellescategories, des données numériques, il faut fixer les types
def read_data(filepath: str):
df = pd.read_csv(filepath, delimiter=";")
return df
def fix_datatypes(df):
cat_columns = ['code_station', 'libelle_station', ...]
for c in cat_columns:
df[c] = df[c].astype('category')
df["date_observation"] = pd.to_datetime(df["date_observation"])
df = read_data("ecoulements.csv")
fix_datatypes(df)Important fixer les types des séries permet de profiter de toutes les fonctionnalités associées (e.g. catégories, temps)
Calculer des statistiques descriptives
Sur une dataframe, on peut :
- calculer des moyennes, sommes, médianes :
df["Age"].median() - agréger avec une liste de fonctions prédéfinies (
min,max,mean, …) ou arbitrairesdf.agg({"Age": ["min", "max", "median", "skew"]})
Les statistiques peuvent être calculées sur des sous-groupes formés par groupby:
titanic = pd.read_csv("data/titanic.csv")
# On peut regrouper les données par catégories et calculer
# des statistiques sur les sous-groupes
titanic = pd.read_csv("titanic.csv")
grouped = titanic[["Sex", "Pclass", "Age", "Fare"]].groupby(["Sex", "Pclass"])
print(grouped.size())
print(grouped.mean()) PassengerId Survived Pclass ... Fare Cabin Embarked
0 1 0 3 ... 7.2500 NaN S
1 2 1 1 ... 71.2833 C85 C
2 3 1 3 ... 7.9250 NaN S
Sex Pclass
female 1 94
2 76
3 144
male 1 122
2 108
3 347
dtype: int64
Age Fare
Sex Pclass
female 1 34.611765 106.125798
2 28.722973 21.970121
3 21.750000 16.118810
male 1 41.281386 67.226127
2 30.740707 19.741782
3 26.507589 12.661633Pivot
Partant d’une représentation empilée (stacked), on peut réarranger les données avec la méthode pivot ou pivot_table (permet d’agréger) :
- index : la ou les colonnes qui servent d’index
- columns : les valeurs que ces colonnes prennent deviennent les colonnes
- values : les valeurs à l’intersection des valeurs d’index et des valeurs de colonnes
df = pd.DataFrame(
{
"A": ["one", "one", "two", "three"] * 6,
"B": ["bidule", "machin", "truc"] * 8,
"C": ["foo", "foo", "foo", "bar", "bar", "bar"] * 4,
"D": np.random.randn(24),
"E": np.random.randn(24),
"F": [datetime.datetime(2013, i, 1) for i in range(1, 13)]
+ [datetime.datetime(2013, i, 15) for i in range(1, 13)],
}
)
print(df)
pivoted = pd.pivot_table(df, values="D", index=["A", "B"], columns=["C"])
print(pivoted)
print(pivoted.loc[("one", "bidule"), "bar"]) A B C D E F
0 one bidule foo 0.029577 0.050984 2013-01-01
1 one machin foo -0.442099 -0.106008 2013-02-01
2 two truc foo 1.953053 1.265677 2013-03-01
3 three bidule bar -1.299982 0.762093 2013-04-01
4 one machin bar 0.652348 -0.323386 2013-05-01
5 one truc bar 1.433064 -1.818466 2013-06-01
6 two bidule foo 2.291547 -0.087507 2013-07-01
7 three machin foo -1.203084 -0.089323 2013-08-01
8 one truc foo 1.022026 -1.088463 2013-09-01
9 one bidule bar -0.433758 2.892209 2013-10-01
...
C bar foo
A B
one bidule 0.243642 -0.317265
machin -0.183342 -0.318656
truc 0.942341 1.991506
three bidule -0.641189 NaN
machin NaN -1.362425
truc -1.088678 NaN
two bidule NaN 1.272628
machin -0.686480 NaN
truc NaN 1.024224
0.24364165912866229pivot retourne une erreur en cas de doublons (moins flexible). pivot_table agrège les doublons (e.g. défaut : aggfunc='mean').
Joindre des tables
On peut joindre plusieurs dataframes par des clés, e.g. :
- une table définit des campagnes de mesures avec leurs dates, l’opérateur, etc…
- une table contient des observations, associées à un identifiant de campagne de mesures
Ces tableaux peuvent être joints, par exemple pour lister les observations réalisées par un opérateur.
pd.merge dans les cheatsheets pandasTracer des données
Pandas est interfacée avec matplotlib pour le tracé d’histogrammes (hist), nuages de points (scatter), séries (plot, même avec le temps !)
Les fonctions de tracé conduisent à des tracés indépendants pour des données groupées groupby.
Tracés avec Matplotlib
Introduction
Matplotlib : Numerical Python
- Librairie python pour le tracé
- offre une multitude de fonctions de tracés plans (line plot, image, histograms), 3D statiques ou animés
Adapting the requirements laid out by John Hunter Matplotlib should:
- Support users of the Scientific Python ecosystem;
- Facilitate interactive data exploration;
- Produce high-quality raster and vector format outputs suitable for publication;
- Provide a simple graphical user interface and support embedding in applications;
- Be understandable and extensible by people familiar with data processing in Python;
- Make common plots easy, and novel or complex visualizations possible.
- Documentation : https://matplotlib.org/doc/stable/,
- Tutoriels : https://lectures.scientific-python.org/intro/matplotlib/index.html
- Tutoriel de N. P. Rougier : https://github.com/rougier/matplotlib-tutorial
- Le livre de N.P. Rougier : Scientific Visualization
- Beyond the basics: Data Visualization in Python by S. Molin
- Les exemples sur https://matplotlib.org/stable/gallery/index
- Code source : https://github.com/matplotlib/matplotlib
Matplotlib for beginners
Matplotlib for intermediate users
Les cheatsheats (1/2)
Les cheatsheats (2/2)
Apprentissage automatique avec scikit-learn
Introduction à l’apprentissage supervisé
Quelques concepts clés pour fixer les idées :
On suppose disposer de données \(x_i\) qui peuvent être étiquetées \(y_i\)
Apprentissage supervisé :
Etant donnés des paires \((x_i, y_i) \sim P_{X, Y}\), apprendre une fonction \(f_\theta\) (un prédicteur) qui minimise le risque réel :
\[ R_r(f) = \int_{X \ times Y} \mathcal{L}(y, f_\theta(x)) p_{X, Y}(x,y) dx dy \]
- Etant données un échantillonnage \((x_i, y_i), i \in [|0, N-1|]\), on peut calculer le risque empirique :
\[ R_e(f) = \sum_{i=0}^{N-1} \mathcal{L}(y_i, f_\theta(x_i)) \]
Question : comment prédire l’étiquette \(y\) associée à des \(x\) étant données des paires d’échantillons \((x_i, y_i)\)
Attention nous ne pourrons pas introduire tout les concepts liés au machine learning
Qu’apporte scikit-learn ? Une vue d’ensemble
Scikit-learn est une librairie python qui offre:
- des intefaces à plusieurs jeux de données classiques sklearn.datasets
- des fonctions pour prétraiter les données sklearn.preprocessing (e.g. normaliser, traiter les données catégorielles, …), de réduction de dimensions sklearn.feature_selection, sklearn.feature_extraction
- différentes familles de prédicteurs : modèles linéaires, arbres, svm,
- voir des aggrégats de prédicteurs
- des métriques d’évaluation sklearn.metrics
- des algorithmes d’évaluation du risque réel cross_validation,
- des algorithmes de recherche des meilleurs hyperparamètres sklearn.model_selection
Le tout étant pipelinable et disposant d’une interface commune.
from sklearn.datasets import load_iris
from sklearn.model_selection import train_test_split
from sklearn.linear_model import LogisticRegression
# Chargement des données
iris = load_iris()
X, y = iris.data, iris.target
# Construction d'un pli d'entrainement et un pli de test
X_train, X_test, y_train, y_test = train_test_split(X, y, test_size=0.2)
# Construction du prédicteur et entrainement
clf = make_pipeline(StandardScaler(), KNeighborsClassifier(3))
clf.fit(X_train, y_train)
# Evaluation du risque sur un pli de test
score = clf.score(X_test, y_test)
# Inférence sur le pli de test
y_test_pred = clf.transform(X_test)Exemples de régression et classification
Ressources

Produced with quarto