4- Traitement d'une grande base de données géolocalisées avec pandas
Environnement virtuel
Pour réaliser les exercices ci-dessous, on pourra se créer un environnement virtuel, par exemple en utilisant uv :
Pour installer uv :
Pour créer un environnement virtuel, par exemple avec python 3.12:
Pour installer les dépendances de ce TP
Premiers pas avec Pandas
Series : une collection indexée de valeurs
Une Series est une structure unidimensionnelle qui peut contenir n'importe quel type de données.
Créer et manipuler une Series
Ouvrez un interpréteur Python ou créez un script test_pandas.py et testez le code suivant :
Les éléments d'une série peuvent être accédés de différentes manières. Une série peut être :
- indexée, slicée comme un tableau numpy avec
iloc[]: accès par position (entier) - indexée, slicée par l'index (\(\approx\) comme un dictionnaire) avec
loc[]: accès par étiquette (label)
Accéder aux éléments d'une Series
Poursuivez votre code avec l'accès aux éléments d'une série :
DataFrame : une table de données
Une DataFrame est une structure bidimensionnelle (tableau) avec des colonnes potentiellement de types différents.
Il est possible créer des dataframes de plusieurs manières :
-
à partir d'un dictionnaire de Series ou l'on spécifie le nom des colonnes et les séries de valeurs associées
-
à partir d'un dictionnaire de listes ou l'on spécifie le nom des colonnes et les listes de valeurs associées, sans spécifier l'index. Dans le cas précédent, des valeurs manquantes (
NaN) seront insérées automatiquement pour les lignes sans valeur. -
à partir d'une liste de dictionnaires, liste qui énumère les lignes de la table
Créer une DataFrame
Construisez une dataframe pandas à partir des éléments du tableau suivant. Vous n'êtes pas obligé de recopier toutes les données, juste 3 ou 4 lignes suffisent pour tester la création de la DataFrame 😊.
| Classe | Rayon (R/R☉) | Masse (M/M☉) | Luminosité (L/L☉) | Température (K) | Etoile |
|---|---|---|---|---|---|
| O2 | 12 | 100 | 800,000 | 50,000 | BI 253 |
| O6 | 9.8 | 35 | 180,000 | 38,000 | Theta¹ Orionis C |
| B0 | 7.4 | 18 | 20,000 | 30,000 | Phi¹ Orionis |
| B5 | 3.8 | 6.5 | 800 | 16,400 | Pi Andromedae A |
| A0 | 2.5 | 3.2 | 80 | 10,800 | Alpha Coronae Borealis A |
| A5 | 1.7 | 2.1 | 20 | 8,620 | Beta Pictoris |
| F0 | 1.3 | 1.7 | 6 | 7,240 | Gamma Virginis |
| F5 | 1.2 | 1.3 | 2.5 | 6,540 | Eta Arietis |
| G0 | 1.05 | 1.10 | 1.26 | 5,920 | Beta Comae Berenices |
| G2 | 1 | 1 | 1 | 5,780 | Sun |
| G5 | 0.93 | 0.93 | 0.79 | 5,610 | Alpha Mensae |
| K0 | 0.85 | 0.78 | 0.40 | 5,240 | 70 Ophiuchi A |
| K5 | 0.74 | 0.69 | 0.16 | 4,410 | 61 Cygni A |
| M0 | 0.51 | 0.60 | 0.072 | 3,800 | Lacaille 8760 |
| M5 | 0.18 | 0.15 | 0.0027 | 3,120 | EZ Aquarii A |
| M8 | 0.11 | 0.08 | 0.0004 | 2,650 | Van Biesbroeck's star |
| L1 | 0.09 | 0.07 | 0.00017 | 2,200 | 2MASS J0523−1403 |
La notation (R/R☉) signifie "Rayon en unités de rayon solaire", de même pour la masse et la luminosité.
Inspection et informations sur une DataFrame
On peut inspecter une DataFrame de plusieurs façons :
head(): affiche les premières lignes (5 par défaut)info(): informations générales sur la structuredescribe(): statistiques descriptives (uniquement colonnes numériques par défaut)shape: tuple (nb_lignes, nb_colonnes)
Inspecter une DataFrame
Créez la DataFrame suivante et explorez ses propriétés :
import pandas as pd
import numpy as np
# Création d'une DataFrame avec des types variés
df = pd.DataFrame({
"station": ["Station A", "Station B", "Station C", "Station D"],
"temperature": [15.5, 18.2, 16.8, 17.3],
"precipitation": [2.5, 0.0, 1.2, np.nan],
"date": ["2024-01-15", "2024-01-16", "2024-01-17", "2024-01-18"]
})
# Afficher la dataframe
print("DataFrame :")
print(df)
print("\n")
# Afficher les premières lignes
print("Premières lignes :")
print(df.head(2))
print("\n")
# Informations sur la structure
print("Informations sur la DataFrame :")
df.info()
print("\n")
# Types des colonnes
print("Types des colonnes :")
print(df.dtypes)
print("\n")
# Statistiques descriptives
print("Statistiques descriptives :")
print(df.describe())
print("\n")
# Noms des colonnes
print(f"Nom des colonnes : {df.columns.tolist()}\n")
# Nombre de lignes et colonnes
print(f"Dimensions : {df.shape[0]} lignes, {df.shape[1]} colonnes\n")
Corriger les types de données
Par défaut, pandas infère les types des colonnes. Cependant, il est souvent nécessaire de les corriger pour bénéficier de toutes les fonctionnalités, en particulier concernant les colonnes catégorielles et les dates.
Eléments de documentation utiles :
astype(): conversion de type,pd.to_datetime(): conversion en datetime (infère automatiquement le format),pd.to_numeric(): conversion en type numérique avec gestion des cas d'erreurs (e.g.errors='coerce'),- Type
category: économise de la mémoire et donne accès à des méthodes spécifiques (.cat.categories), describe(include='category'): statistiques pour les colonnes catégorielles
Conversion de types
Reprenez la DataFrame de l'exercice 3 et corrigez les types. Prenez le temps d'observer les nouvelles fonctionnalités disponibles après conversion : statistiques descriptives pour les colonnes catégorielles et dates.
import pandas as pd
df = pd.DataFrame({
"station": ["Station A", "Station B", "Station B", "Station D"],
"temperature": [15.5, 18.2, 16.8, 17.3],
"precipitation": [2.5, 0.0, 1.2, np.nan],
"date": ["2024-01-15", "2024-01-16", "2024-01-17", "2024-01-18"],
"qualite": ["bon", "moyen", "bon", "excellent"]
})
print("Types avant conversion :")
print(df.dtypes)
print()
# Conversion de la colonne 'station' en type category
df['station'] = df['station'].astype('category')
# Conversion de la colonne 'qualite' en type category
df['qualite'] = df['qualite'].astype('category')
# Conversion de la colonne 'date' en datetime
df['date'] = pd.to_datetime(df['date'])
print("Types après conversion :")
print(df.dtypes)
print()
# Pour les colonnes catégorielles, on peut accéder aux catégories
print("Catégories de 'qualite' :", df['qualite'].cat.categories.tolist())
# Les statistiques descriptives changent !
print("Statistiques sur les catégories :")
print(df.describe(include='category'))
print("Statistiques sur les dates :")
print(df[['date']].describe())
Sélectionner et filtrer des données
La sélection et le filtrage des données dans une DataFrame peuvent se faire de plusieurs manières :
df['colonne']: sélection d'une colonne (retourne une Series)df[['col1', 'col2']]: sélection de plusieurs colonnes (retourne une DataFrame)df[condition]: filtrage par condition booléenne&(ET),|(OU) : combinaison de conditions (les parenthèses sont obligatoires !)
Sélection de colonnes et de lignes
import pandas as pd
df = pd.DataFrame({
"station": ["A", "B", "C", "D", "E"],
"temperature": [15.5, 18.2, 16.8, 17.3, 14.9],
"precipitation": [2.5, 0.0, 1.2, 3.8, 5.1],
})
# Sélectionner une colonne (retourne une Series)
print("Colonne 'temperature' :")
print(df['temperature'])
print(type(df['temperature']))
print()
# Sélectionner plusieurs colonnes (retourne une DataFrame)
print("Colonnes 'station' et 'temperature' :")
print(df[['station', 'temperature']])
print()
# Sélectionner des lignes par position avec iloc
# avec la possibilité de slicer
print("Lignes 1 à 3 (position) :")
print(df.iloc[1:4])
print()
# Filtrer les lignes selon une condition
print("Stations avec température > 16 :")
print(df[df['temperature'] > 16])
print()
# Filtrer avec plusieurs conditions
print("Stations avec température > 16 ET precipitation < 2 :")
print(df[(df['temperature'] > 16) & (df['precipitation'] < 2)])
Indexation par étiquette avec set_index
Une fonctionnalité puissante de pandas est la possibilité de changer l'index d'une DataFrame pour utiliser une ou plusieurs colonnes comme index. Cela permet un accès plus intuitif aux données en utilisant l'indexation par étiquettes avec loc[].
set_index(): définit une ou plusieurs colonnes comme indexreset_index(): remet l'index numérique par défaut- Avec un index personnalisé,
loc[]devient très puissant pour sélectionner des sous-ensembles
Changer l'index d'une DataFrame
import pandas as pd
df = pd.DataFrame({
"code_station": ["A01", "B02", "A01", "C03", "B02"],
"date": ["2024-01-15", "2024-01-15", "2024-01-16", "2024-01-15", "2024-01-16"],
"debit": [10.5, 8.2, 11.3, 15.4, 7.9]
})
print("DataFrame originale :")
print(df)
print()
# Changer l'index pour utiliser code_station
df_indexed = df.set_index('code_station')
print("Après set_index('code_station') :")
print(df_indexed)
print()
# Accéder à toutes les données de la station A01
print("Toutes les observations de la station A01 :")
print(df_indexed.loc['A01'])
print()
# Revenir à l'index numérique par défaut
df_reset = df_indexed.reset_index()
print("Après reset_index() :")
print(df_reset)
Ajouter et supprimer des colonnes/lignes
On peut ajouter dynamiquement des colonnes dans une DataFrame en assignant une liste ou une Series à une nouvelle colonne. Pour supprimer des colonnes ou des lignes, on utilise la méthode drop().
- Ajout de colonne :
df['nouvelle_col'] = valeurs drop(): suppression de lignes (axis=0) ou colonnes (axis=1)inplace=True: modifie la DataFrame directement (sinon retourne une copie)
Manipuler la structure de la DataFrame
import pandas as pd
df = pd.DataFrame({
"station": ["A", "B", "C"],
"temperature": [15.5, 18.2, 16.8],
"precipitation": [2.5, 0.0, 1.2]
})
print("DataFrame initiale :")
print(df)
print()
# Ajouter une nouvelle colonne
df['humidite'] = [65, 70, 68]
print("Après ajout de la colonne 'humidite' :")
print(df)
print()
# Ajouter une colonne calculée
df['t_fahrenheit'] = df['temperature'] * 9/5 + 32
print("Après ajout de la température en Fahrenheit :")
print(df)
print()
# Supprimer une colonne
df_sans_f = df.drop('t_fahrenheit', axis=1)
print("Après suppression de 't_fahrenheit' (axis=1 pour colonnes) :")
print(df_sans_f)
print()
# Supprimer des lignes (par index)
df_sans_ligne = df.drop(1, axis=0)
print("Après suppression de la ligne d'index 1 (axis=0 pour lignes) :")
print(df_sans_ligne)
Gérer les valeurs manquantes
Nous avons vu que la méthode info() permet de compter le nombre de valeurs manquantes par colonne. On peut avoir besoin de détecter, supprimer ou remplacer ces valeurs manquantes (NaN).
isna(): détecte les valeurs manquantesdropna(): supprime les lignes/colonnes avec NaNfillna(): permet d'imputer une valeur aux valeurs manquantes
Traiter les données manquantes
import pandas as pd
import numpy as np
df = pd.DataFrame({
"station": ["A", "B", "C", "D", "E"],
"temperature": [15.5, np.nan, 16.8, 17.3, np.nan],
"precipitation": [2.5, 0.0, np.nan, 3.8, 5.1]
})
print("DataFrame avec valeurs manquantes :")
print(df)
print()
# Détecter les valeurs manquantes
print("Valeurs manquantes (True = manquant) :")
print(df.isna())
print()
# Compter les valeurs manquantes par colonne
print("Nombre de valeurs manquantes par colonne :")
print(df.isna().sum())
print()
# Supprimer les lignes avec des valeurs manquantes
df_sans_na = df.dropna()
print("Après dropna() (suppression des lignes avec NaN) :")
print(df_sans_na)
print()
# Remplir les valeurs manquantes
df_filled = df.fillna(0)
print("Après fillna(0) (remplacement des NaN par 0) :")
print(df_filled)
print()
# Remplir avec la moyenne de la colonne
df_mean = df.copy()
df_mean['temperature'] = df_mean['temperature'].fillna(df_mean['temperature'].mean())
print("Après remplacement des NaN de température par la moyenne :")
print(df_mean)
Grouper et agréger des données
Pandas permet de calculer facilement des statistiques sur des groupes de données :
groupby(): regroupe les données selon une ou plusieurs colonnesagg(): applique une ou plusieurs fonctions d'agrégation- Fonctions courantes :
mean(),sum(),count(),min(),max(),std()
Utiliser groupby
import pandas as pd
df = pd.DataFrame({
"region": ["Grand-Est", "Grand-Est", "Nouvelle-Aquitaine",
"Nouvelle-Aquitaine", "Grand-Est"],
"station": ["A", "B", "C", "D", "E"],
"temperature": [15.5, 18.2, 20.1, 19.5, 16.3],
"precipitation": [2.5, 0.0, 1.2, 3.8, 5.1]
})
print("DataFrame :")
print(df)
print()
# Grouper par région et calculer la moyenne des températures
grouped = df.groupby('region')
print("Moyennes par région :")
print(grouped[["temperature", "precipitation"]].mean())
print()
# Compter le nombre de stations par région
print("Nombre de stations par région :")
print(grouped.size())
print()
# Appliquer plusieurs fonctions d'agrégation
print("Statistiques détaillées par région :")
print(grouped['temperature'].agg(['mean', 'min', 'max', 'std']))
Réarranger des données entre format large et format long
Pandas offre des méthodes pour réarranger les données entre des formats "long" (en Anglais long ou stacked) et "large" (en Anglais: wide ou record). Le format "long", empilé, correspond à une table où chaque ligne est une observation unique, essentiellement avec un index qui revient à être un identifiant correspondant au numéro de la donnée.
Mais c'est avec le format large que l'index prends toute son importance. On passe d'un format long à un format en définissant :
- une ou plusieurs colonnes comme index
- une ou plusieurs colonnes comme colonnes
- une ou plusieurs colonnes comme valeurs
Cette manipulation est illustrée sur la documentation pandas/Reshaping and pivot tables:
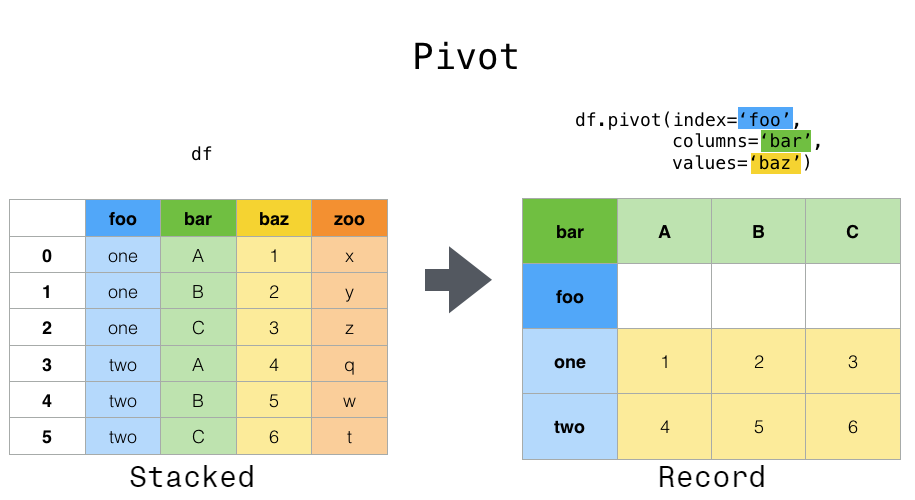
Sur cette image, on voit, après l'application du pivot que les mêmes données sont réarrangées pour que :
- verticalement, l'index correspond aux valeurs prises par la colonne
foo - horizontalement, les colonnes correspondent aux valeurs prises par la colonne
bar - les valeurs de la table correspondent aux valeurs prises par la colonne
baz
Il peut y avoir des doublons à la croisée index/columns. Dans ce cas, la méthode pivot retourne une erreur quand la méthode pivot_table agrège les doublons.
Regardons un exemple. Imaginons que nous avons collecté des données de température dans différentes pièces d'une maison, à six dates différentes, toutes ces mesures étant regroupées dans une table.
df = pd.DataFrame(
{
"pièce": ["chambre", "salon", "cuisine"] * 6,
"température": [20, 22, 21, 21, 23, 22, 19, 21, 20, 22, 24, 23, 20, 22, 21, 21, 23, 22],
"date": [datetime.datetime(2025, 7, 25)]*3 + [datetime.datetime(2025, 7, 26)]*3 + [datetime.datetime(2025, 7, 27)]*3 +
[datetime.datetime(2025, 7, 28)]*3 + [datetime.datetime(2025, 7, 29)]*3 + [datetime.datetime(2025, 7, 30)]*3,
}
)
print(df)
On aimerait calculer des statistiques de température par pièce, ou simplement afficher la température pour chacune des pièces au cours du temps. On pourrait bien sûr masquer la colonne pièce et faire des filtres successifs, mais il est plus simple de réarranger les données pour que chaque pièce corresponde à une colonne. On peut passer sur un format large en utilisant la méthode pivot_table() :
pivoted = pd.pivot_table(df, values="température", index=["date"], columns=["pièce"])
print(pivoted)
De cette manière, on accède plus facilement aux données en utilisant l'indexation par étiquette pour répondre à des questions du type : "Quelles sont les températures, au cours du temps, dans la cuisine ?"
Et on peut même facilement tracer ces valeurs :
import matplotlib.pyplot as plt
pivoted["chambre"].plot(title="Température de la chambre au fil du temps")
plt.xlabel("Date")
plt.ylabel("Température (°C)")
plt.show()
Et en fait, vous pourriez même tracer toutes les pièces en une seule commande :
pivoted.plot(title="Température de la chambre au fil du temps")
plt.xlabel("Date")
plt.ylabel("Température (°C)")
plt.show()
Tracer de la séquence principale
Nous allons réaliser un petit exercice de mise en oeuvre de pandas pour tracer le diagramme de Hertzsprung–Russell qui fait en particulier apparaître la "Séquence principale", une relation qui lit la luminosité d'une étoile en fonction de sa température de surface. Cet exercice est inspiré du travail de Eduardo Martin Calleja.
Pour ce faire, nous utilisons les données du catalogue Hipparcos de l'équipe VizieR qui contient des données pour plus de 100 000 étoiles. Un extrait de quelques colonnes est disponible au format csv ici : hipparcos.csv.
Tracer la séquence principale
En utilisant pandas et matplotlib, écrivez un script python qui :
- charge les données du fichier CSV dans une DataFrame pandas (voir la fonction pandas.read_csv). Prenez le temps de regarder le contenu de ce fichier, en particulier le séparateur, le fait qu'il y a des commentaires, ...
- inspecte les données pour comprendre les colonnes disponibles,
- calcule la magnitude absolue \(M_v\) à partir de la parallaxe \(p\) (en milliarcsecondes) et de la magnitude apparente \(V_{mag}\) en utilisant la formule ci-dessous. La parallale permet de calculer la distance de l'étoile.
- calcule la luminosité \(L\) en unités solaires à partir de la magnitude absolue \(M_v\) en utilisant la formule :
- trace la luminosité relative \(L\) en fonction de l'index de couleur \(B-V\) (voir la fonction pandas.DataFrame.plot.scatter).
Cela devrait produire un graphique similaire :

Sur ce graphique, la diagonale un peu courbe porte le nom de "séquence principale".
Warning
Je ne suis pas tout à fait sûr des formules. Une lecture par une personne experte en astrophysique serait bienvenue pour valider ou corriger les formules utilisées ici ! 🧐
Utilisation de pandas pour explorer des données sur les écoulements des rivières
Nous avons fait le tour d'un certain nombre de fonctionnalités de base de pandas que nous allons désormais appliquer sur des données d'écoulement des rivières.
Pour poursuivre notre travail sur Pandas, nous allons utiliser un export CSV de l'API Hubeau sur l'écoulement des cours d'eau pour l'année 2024, dans la région Grand-Est (code INSEE 44), et plus particulièrement le département de Meurthe et Moselle (Code INSEE 54) : ecoulements.csv.
Chargement et inspection des données
Question
En utilisant la documentation pandas, votre objectif ici est d'écrire un script python utilisant pandas pour répondre aux questions suivantes :
- chargez ces données au format CSV dans une dataframe pandas. Pensez à inspecter ce fichier, le séparateur est peut-être un peu particulier, la première ligne contient peut-être les noms des colonnes. La fonction pandas.read_csv pourrait être utile,
- quel est le nombre de lignes de données ?
- quel est le nombre de colonnes ? Quels sont les intitulés de ces colonnes ? Quels sont les types de ces colonnes ?
Warning
Conseil de mise en oeuvre
Je vous conseille de coder une fonction par question et d'invoquer ces fonctions dans le bloc conditionnel if __name__ == '__main__':. Votre script python pourra par exemple avoir la structure suivante :
# coding: utf-8
# Standard imports
import logging
# External imports
import pandas as pd
def read_data(filepath: str):
...
return df
def nombre_lignes():
df = read_data(...)
num_lignes = ...
print(f"La dataframe pandas contient {num_lignes} lignes")
if __name__ == '__main__':
nombre_lignes()
Je vous invite à suivre cette même approche pour les prochaines questions pour garder un code lisible.
Correction des types des colonnes
Certaines (beaucoup) colonnes sont du type générique object et comme indiqué dans la documentation https://pandas.pydata.org/docs/user_guide/basics.html#dtypes,
arbitrary objects may be stored using the object dtype, but should be avoided to the extent possible (for performance and interoperability with other libraries and methods).
Vous avez dû constater précédemment que plusieurs colonnes, de différentes natures, sont affectées du type pandas object. Pour pouvoir manipuler convenablement les données et profiter pleinement de toutes les opérations applicables, il est nécessaire d'indiquer à pandas le bon type de ces colonnes.
Question
Vous devez écrire une fonction fix_datatypes(df) qui applique les changements de type suivants pour arriver au résultat ci-dessous :
code_station category
libelle_station string[python]
uri_station string[python]
code_departement category
libelle_departement string[python]
code_commune category
libelle_commune string[python]
code_region category
libelle_region string[python]
code_bassin category
libelle_bassin string[python]
coordonnee_x_station float64
coordonnee_y_station float64
code_projection_station category
libelle_projection_station string[python]
code_cours_eau category
libelle_cours_eau string[python]
uri_cours_eau string[python]
code_campagne category
code_reseau category
libelle_reseau string[python]
uri_reseau string[python]
date_observation datetime64[ns]
code_ecoulement category
libelle_ecoulement string[python]
latitude float64
longitude float64
Quelques indications de mise en oeuvre :
- Pour changer le type d'une colonne en
string, si la colonne s'appelle cname :df[cname] = df[cname].astype('string'), - pour changer le type d'une colonne en
category, si la colonne s'appelle cname :df[cname] = df[cname].astype('category') - pour changer le type d'une colonne en
datetime, pandas offre la fonction to_datetime qui va, par défaut, essayer d'inférer le format de la date,
Note: il est possible d'accéder à la liste des catégories d'une colonne catégorielle par df[cname].cat.categories
L'un des bienfaits d'avoir corriger ces types de colonnes est le résultat de l'appel à la fonction pandas.DataFrame.describe.
Question
Comparez la sortie de la fonction describe appliquée sur votre DataFrame avant et après avoir corrigé le type des colonnes.
Warning
Attention, comme vous pourrez le vérifier, la méthode describe s'applique par défaut sur les données numériques et de type object. Lorsque des colonnes ont correctement spécifiées comme de type catégoriel, il faut spécifiquement invoquer la méthode describe avec l'argument include='category'
- Qu'observez-vous sur les colonnes catégorielles ? Qu'observez-vous sur les statistiques descriptives avant et après conversion du type ?
- Qu'observez-vous sur la colonne des dates d'observation ? Quelle est l'étendue temporelle de la collecte des données ?
- Combien y a-t-il de codes région uniques ? Combien y a-t-il de codes commune uniques ?
- Quel est le code d'écoulement le plus présent ? Vous trouverez la signification de cet écoulement sur la documentation de l'observatoire des étiages
Accès aux lignes par position ou par étiquette
Pour accéder aux lignes, vous pouvez utiliser une indexation par position ou une indexation par étiquette.
Tout d'abord, commençons par l'accès indexé par position :
Question
- Quelle est la vingtième donnée ? iloc pourrait être utile. Vous trouvez bien une mesure au ruisseau des templiers à Flin ?
Une fonctionnalité très puissante de pandas est de pouvoir modifier la donnée utilisée pour indexer les tables pandas. Lorsqu'une dataframe est construite, elle dispose d'un index entier, qui correspond au numéro de ligne de la donnée. En d'autres termes, les deux appels ci-dessous retourneraient le même résultat :
Par contre, si vous changez l'indexation pour que l'index soit maintenant la liste des code_station :
Il devient possible d'indexer la table par une valeur de code station :
Le résultat est une dataframe pandas avec toutes les données pour lesquelles la colonne code_station est "B2030001". Vous pouvez revenir à l'indexation par défaut grâce à reset_index :
Ici, par l'appel de reset_index, subdf retrouve l'indexation par défaut.
Question
On voudrait trier les observations par le type d'écoulement.
- Commencez par identifier les valeurs catégorielles que peut prendre la colonne "code_ecoulement". Peut être que pandas.Series.cat.categories peut être utile ?
- Combien y a-t-il d'observations pour lesquelles les écoulements sont de code 1f, correspondant au libellé "Ecoulement visible faible" ? Utilisez
set_indexetlocà bon escient ! - Quelle est la station pour laquelle il y a le plus d'observations avec un code d'écoulement 1f ? (Vous vous souvenez de
describe?)
Suppression de lignes et de colonnes
Pour finir cette première partie, on va également voir comment supprimer des lignes, des colonnes, etc... Pour supprimer des lignes et/ou des colonnes, il suffit d'invoquer la méthode drop d'une dataframe en spécifiant axis=0 pour les lignes, axis=1 pour les colonnes. Cette méthode peut modifier une dataframe "en place" en spécifiant inplace=True.
Par exemple :
# Suppression des entrées d'une station : on utilise l'astuce de l'indexation par étiquette !
df = df.set_index('code_station')
df.drop("B2030001", axis=0, inplace=True)
# Suppression de quelques colonnes
df.drop(["code_projection_station", "libelle_projection_station"], axis=1)
Question
Construisez une nouvelle dataframe en :
- excluant les colonnes 'code_departement', 'libelle_departement', 'code_commune', 'libelle_commune', 'code_region', 'libelle_region', 'code_bassin', 'libelle_bassin', 'code_campagne', 'code_reseau', 'libelle_reseau', 'uri_reseau'
- excluant les lignes dont le code d'écoulement est 2 ou 3
Localiser des données
Pour terminer cette partie sur le travail des données d'écoulement, on se propose d'exploiter la géolocalisation des stations. On souhaite représenter les stations de collecte sur une carte (plane).
Il n'existe pas d'unique système de géo-référencement à la surface du globe. Le lecteur intéressé pourra par exemple consulter ce cours sur les systèmes de coordonnées de références.
Les données fournies par Hubeau sont géolocalisées.
Question
- Quelles colonnes vous semblent contenir des coordonnées associées aux observations ? (indice : il y en a 4)
- Les colonnes
coordonnee_x_stationetcoordonnee_y_stationsont associées à une projection. Ces projections ont un code et un libellé, quels sont-ils ? - À partir de la documentation http://id.eaufrance.fr/nsa/22, retrouvez-vous le nom de la projection ?
Les systèmes de projection ont des noms (e.g. Lambert 93, WGS84) et également un code EPSG pour les identifier. A la date d'écriture de ce sujet, les coordonnées de notre dataframe sont fournies RGF93 / Lambert 93, de code EPSG 2154. C'est un système de référencement très utilisé en France parce qu'il ne déforme pas trop les angles ni les formes aux latitudes/longitudes de la France Métropolitaine (ce n'est plus vrai dans les DOM/TOM !).
Pour utiliser un géo-référencement dans le tracé de données, il suffit de spécifier la projection à utiliser pour l'axe ainsi que la projection associée aux données tracées comme illustré sur le code ci-dessous.
Example
import matplotlib.pyplot as plt
import cartopy
import cartopy.crs as ccrs
plt.figure(figsize=(15, 15))
crs = ccrs.epsg(9794) # RGF93/Lambert93 v2b
# crs = ccrs.Mercator()
# On définit un axe en spécifiant le système de projection que l'on souhaite utiliser
ax = plt.axes(projection=crs)
# On ajoute quelques éléments graphiques fournit par cartopy
ax.add_feature(cartopy.feature.LAND)
ax.add_feature(cartopy.feature.BORDERS)
ax.coastlines()
# Show the longitude/latitude lines
ax.gridlines(draw_labels=True)
# On trace des points en spécifiant le système de projection dans lequel les coordonnées
# sont définies. Matplotlib va les convertir dans la projection utilisée par l'axe
cities = [
{"lon": -3.7026, "lat": 40.4165, "name": "Madrid"},
{"lon": 4.3499, "lat": 50.8467, "name": "Bruxelles"},
{"lon": 2.33333, "lat": 48.866667, "name": "Paris"},
{"lon": 6.166667, "lat": 49.133333, "name": "Metz"},
{"lon": 6.2, "lat": 48.6936111, "name": "Nancy"},
{"lon": 4.023584, "lat": 49.24875, "name": "Reims"},
{"lon": 4.07744, "lat": 48.2956, "name": "Troyes"},
{"lon": 7.752222, "lat": 48.573333, "name": "Strasbourg"}
]
ax.scatter([city["lon"] for city in cities],
[city["lat"] for city in cities],
color='blue', linewidth=2, marker='x',
transform=ccrs.Geodetic(),
)
for city in cities:
plt.text(city["lon"]+0.05, city["lat"] , city["name"],
horizontalalignment='left',
transform=ccrs.Geodetic())
plt.show()
Le code ci-dessus donne les images ci-dessous avec une projection pour l'axe Mercator ou RGF93/Lambert 93 v2b.


Question
Produisez une carte des stations de mesure des écoulements. Vous pouvez bien sûr vous inspirer du code ci-dessus.
Note
Pour la mise en oeuvre, vous pouvez probablement ne conserver que les colonnes 'coordonnee_x_station' et 'coordonnee_y_station' et supprimer les doublons
A titre d'exemple, les stations des mesures des observations d'écoulement, dans la région Grand-Est pour l'année 2023 sont localisées sur la carte ci-dessous.

Connexion à l'API Hubeau
Danger
Dans cette partie du TP, lorsque vous utilisez l'API Hubeau, nous utilisons un cache local pour les données; Une fois téléchargées, les données sont stockées localement dans le répertoire d'exécution. Cela permet de limiter le nombre de requêtes réseaux pour récupérer les données, diminue le temps d'exécution de vos programmes mais peut prendre un peu de place sur disque. Je vous invite, une fois le TP terminé, de supprimer ces données :
Jusqu'à maintenant, vous avez travaillé avec un export CSV des données Naïades sur les écoulements. D'ailleurs, la base Naïades contient des données de différentes natures, pas uniquement des données sur l'écoloulement des rivivèes (voir le manuel d'utilisation de Naïades:
- hydrobiologie : poissons, macroinvertébrés, diatomées et macrophytes de 1971 à 2018 avec plus de 3M de taxons répertoriés,
- hydromorphologie : mesures sur la morphologie des rivières avec de l'ordre de 500K transects de 2008 à 2018,
- physicochimie : résultats d'analyses de différents paramètres avec environ 125M d'analyse de 1960 à 2019,
- température : environ 30M de mesures de 2006 à 2020,
- etc.
Pour chacune de ces thématiques, on récupère un certain nombre de fichiers qui contiennent par exemple des informations sur les stations de mesure, les données mesurées, etc. .
Pour faciliter l'accès et l'utilisation des données Naïades, le BRGM a développé une API Rest : Hub'eau. Cette API se présente sous la forme de serveurs vers lequel on peut émettre des requêtes pour obtenir une partie de la base de données filtrée selon certains critères comme : les départements, régions, communes concernées, les périodes de temps des mesures, etc..
On vous fournit le script hubeau.py qui permet d'interface du code python avec quelques endpoints de l'API Hub'Eau, à savoir pour le moment :
- l'API sur les écoulements des cours d'eau https://hubeau.eaufrance.fr/page/api-ecoulement,
- l'API sur l'hydrobiologie https://hubeau.eaufrance.fr/page/api-hydrobiologie,
- l'API sur la température des cours d'eau https://hubeau.eaufrance.fr/page/api-temperature-continu,
- l'API sur la qualité des cours d'eau https://hubeau.eaufrance.fr/page/api-qualite-cours-deau,
- l'API sur le recensement des poissons lors d'opérations de pêches scientifiques à l'électricité https://hubeau.eaufrance.fr/page/api-poisson.
Tip
Si vous voulez contribuer à l'écriture de code, n'hésitez pas à soumettre des pull requests. Le script hubeau.py est mis à disposition sous license CC BY-SA 4.0
Nous utiliserons également ces données durant les deux prochains TPs. Dans ce TP, on ne va s'intéresser qu'à la prise en main de ce code pour s'interfacer avec l'API.
Pour chaque API, vous disposez d'une classe avec une méthode par endpoint, comme illustré sur les exemples ci-dessous :
Example
# API sur les écoulements
ecoulement = hubeau.Ecoulement()
ecoulement.stations(year=2023, code_departement=[54])
ecoulement.observations(year=2023, code_departement=[54])
ecoulement.campagnes(year=2023, code_departement=[54])
# API pour l'hydrobiologie
hydrobio_client = hubeau.Hydrobiologie()
hydrobio_client.indices(year=2023, code_departement=[54])
hydrobio_client.stations_hydrobio(year=2023, code_departement=[54])
hydrobio_client.taxons(year=2023, code_departement=[54])
# API pour la température des cours d'eau
temperature_client = hubeau.Temperature()
temperature_client.chronique(year=2023, code_departement=[54])
temperature_client.station(year=2023, code_departement=[54])
# API sur la qualité des cours d'eau
qualite = hubeau.QualiteCoursEau()
# analyse = qualite.analyse_pc(year=2023, code_departement=[54])
qualite.condition_environnementale_pc(year=2023, code_departement=[54])
qualite.operation_pc(year=2023, code_departement=[54])
qualite.station_pc(year=2023, code_departement=[54])
# API sur les
poisson_client = hubeau.Poisson()
poisson_client.indicateurs(year=2023, code_departement=[54])
poisson_client.observations(year=2023, code_departement=[54])
poisson_client.operations(year=2023, code_departement=[54])
poisson_client.stations(year=2023, code_departement=[54])
Chacun des appels aux endpoints vous retourne une dataframe pandas !
Question
Remplacez la lecture des données d'écoulement, pour le moment réalisée avec le fichier CSV que je vous ai fourni, avec un appel à l'API d'écoulement, endpoint observations.
Récupérez et tracez la carte des coordonnées des observations des écoulements de l'année 2024, région Nouvelle Aquitaine (Code INSEE de région 75)
Solutions
Les solutions sont disponibles ci-dessous :
References
- Data wrangling with pandas
- Maxime Ryckewaert and Diego Marcos and Christophe Botella and Maximilien Servajean and Pierre Bonnet and Alexis Joly 2025. "Applying the maximum entropy principle to neural networks enhances multi-species distribution models", https://doi.org/10.48550/arXiv.2412.19217,
- Elith, Jane, Catherine Graham, Roozbeh Valavi, Meinrad Abegg, Caroline Bruce, Simon Ferrier, Andrew Ford, et al. 2020. "Presence-Only and Presence-Absence Data for Comparing Species Distribution Modeling Methods". Biodiversity Informatics 15 (2): 69-80. https://doi.org/10.17161/bi.v15i2.13384.
- Hands on Data analysis with Pandas - Stéfanie Molin
- Les systèmes de coordonnées de références
- Indices de biodiversité
- API naiades hubeau
- Hydroécorégions de niveau 1
- Euroscipy
- Tutoriel de S. Molin
- Euroscipy 2023